**标题:**Differential gene expression analysis tools exhibit substandard performance for long non-coding RNA-sequencing data DOI(url): https://doi.org/10.1186/s13059-018-1466-5 **发表日期:**24 July 2018 关键词: lncRNA, Differential gene expression, RNA-seq, differential expression 这篇文章详细的分析了不同标准化和差异分析方法在 lncRNA 分析中的差别。一共使用了 25 个分析流程,主要关注点是 lncRNA 和一些低表达 mRNA。使用 15 种指标来评估差异基因的分析方法和标准化方法,一共使用了 6 中不同 RNA-seq 数据集,同时还提供了一个 shiny 网页可视化工具用来展示这些分析结果。按道理类似类型的文章应该达不到这个水平的杂志,lncRNA 分析方法的测评能够发到 Genome Biology 上也是牛,想必定有过人之处。 简单说最后的结论是使用 limma 和 SAMSeq 分析 lncRNA 或者表达量很低的 mRNA 效果要稍微好些,值得注意的是,为了获得至少 50% 的 sensitivity,在实际环境(如临床癌症研究)中研究表达水平时需要超过 80 个样本(what ?)。测试使用的大约一半的方法显示出过多的假阳性,非常不可靠。 lncRNA 研究的主要问题就是表达量太低,在一些软件中往往是要求去除掉表达量很低的基因,这个时候就非常尴尬。 作者为此挑选了一些引用率较高的软件,这些软件的共同点是都有 R 包可以使用,而且都是用原始的 read counts 作为输出。在数据方面,作者使用了不同规模的 6 个数据,基本上可以概括进行差异分析的不同情况。用作者的话说:据我们所知,我们的研究是迄今为止所进行的最大的实例 评估,包括所使用的真实数据集的数量,评估的指标数量以及 DE 流程数量。 使用的差异分析工具如下: 结论是除了 quantile normalization (QN),其它几种标准化的方法都差不多。 另外,终于在文献里看到了 upsetR 画出来的图。 主要检查指标: 这里的分析结果是使用聚类方法展示的,如果看懂了就很能说明问题。 下图中的 abcd 分别代表: a fraction of significantly differentially expressed (SDE) genes detected at 5% FDR, b overlap among pipelines in detecting SDE genes at 5% FDR, c gene ranking agreement, and d similarity of log fold-change (LFC) estimates) DESeq, baySeq, limmaQN,和 NOISeq 聚在一起,总之就是比较差。 QuasiSeq (both settings), edgeR robust (with both tested prior degrees of freedom), limmaVoom+QW, PoissonSeq 和 SAMSeq 相对而言就都比价不错。 文章后面有介绍了一些其它数据的比价结果就不一一详细罗列,最后的建议是:**limma (with variance stabilizing transformation; voom with or without quality weighting; trend) and SAMSeq control the actual FDR reasonably well, while not sacrificing sensitivity。**另外,就是如果做 lncRNA,多送一些样本吧。 文章上传了分析代码,所有几个主要附件都是网页格式,展示所有评测结果还做了个网站。 最后一点让我好奇的是,文章从投稿到接受一共花了 8 个月的时间,这期间都发生了什么,review 又给了哪些意见,什么数据是在 review 下补的 … 本文作者:思考问题的熊 版权声明:本博客所有文章除特别声明外,均采用 知识共享署名-非商业性使用-禁止演绎 4.0 国际许可协议 (CC BY-NC-ND 4.0) 进行许可。 如果你对这篇文章感兴趣,欢迎通过邮箱订阅我的 「熊言熊语」会员通讯,我将第一时间与你分享肿瘤生物医药领域最新行业研究进展和我的所思所学所想,点此链接即可进行免费订阅。文献信息
文献概述
笔记
差异分析工具
Tool (package version)
Pipelines
Citationsa
edgeR (3.14.0)
(1) Exact test based on NB distribution, (2) GLM with NB family, (3) QL, (4–7) robust GLM with four different prior DF
5406
DESeq (1.24.0)
(1) Default, exact test based on NB distribution
4655
DESeq2(1.12.4)
Fits GLM with NB family. (1) Default, (2) independent filtering disabled (setting1), (3) independent filtering disabled and outlier-detection off (setting2)
1364
limma (3.25.21)
Fits linear models on log-transformed counts. (1) Voom, (2) voom (robust), (3) trended, (4) trended (robust), (5) voom+QW, (6) limmaVST, (7) limmaQN
1828
NOISeq (2.12.1)
(1) Default, data-adaptive and non-parametric method
524
baySeq (2.6.0)
(1) Default, Bayesian methods with empirical prior distributions
315
SAMSeq (samr, 2.0)
(1) Default, non-parametric method based on Wilcoxon rank sum statistic
140
PoissonSeq (1.1.2)
(1) Default, uses poisson log-linear model
92
QuasiSeq (1.0.8)
Fits GLM with NB family. (1) QL, (2) QLShrink, (3) QLSPline
57
分析过程
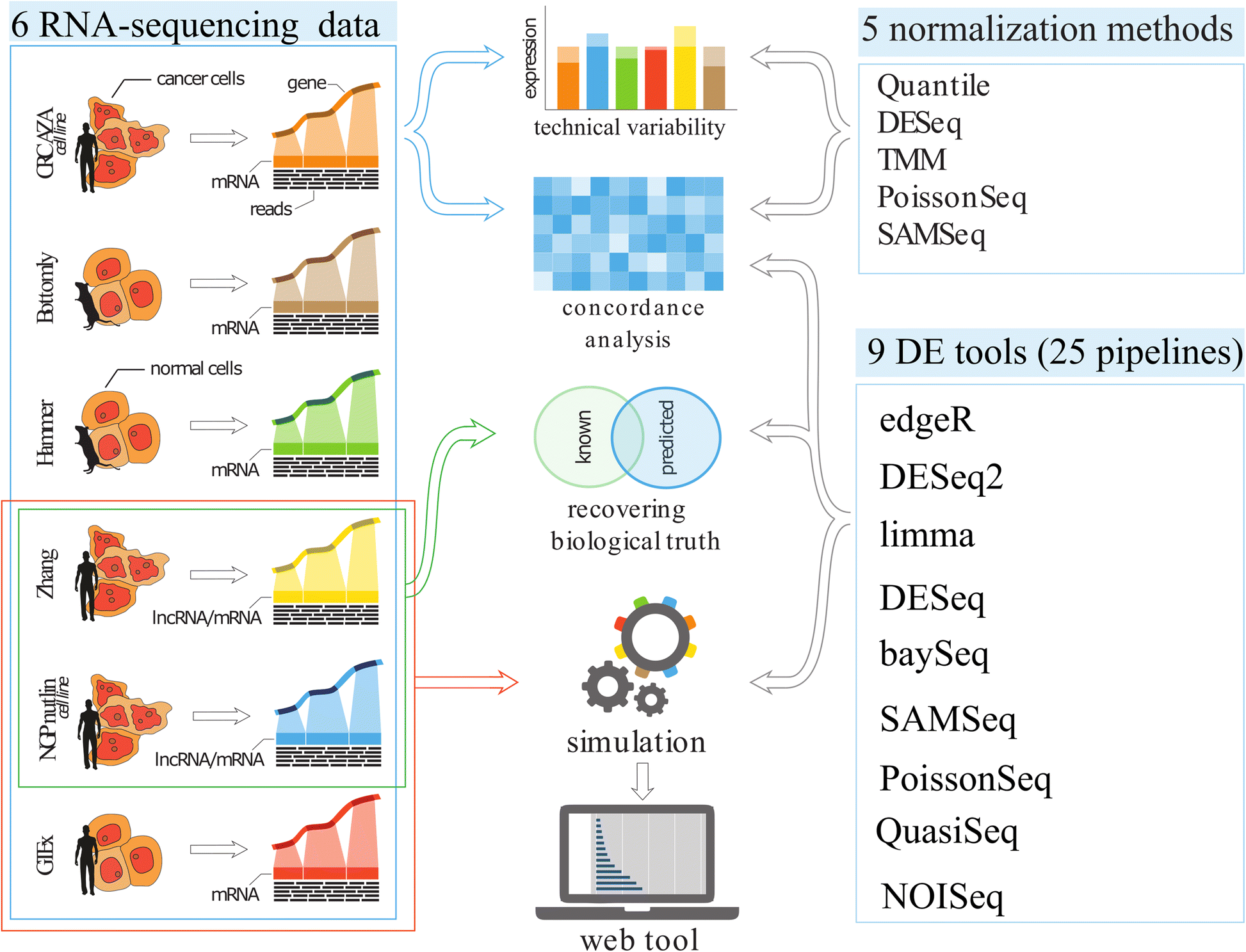
标准化方法比较
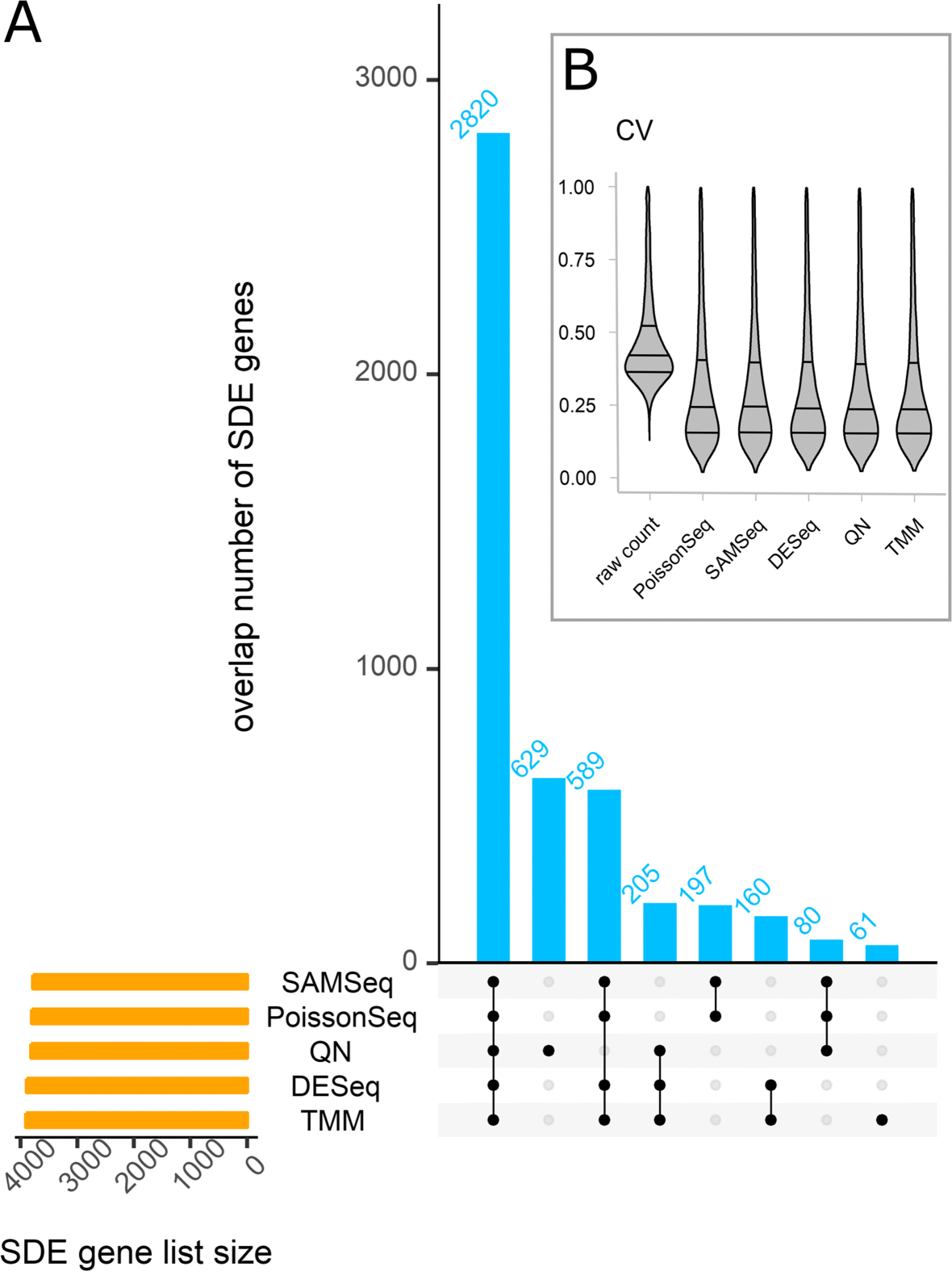
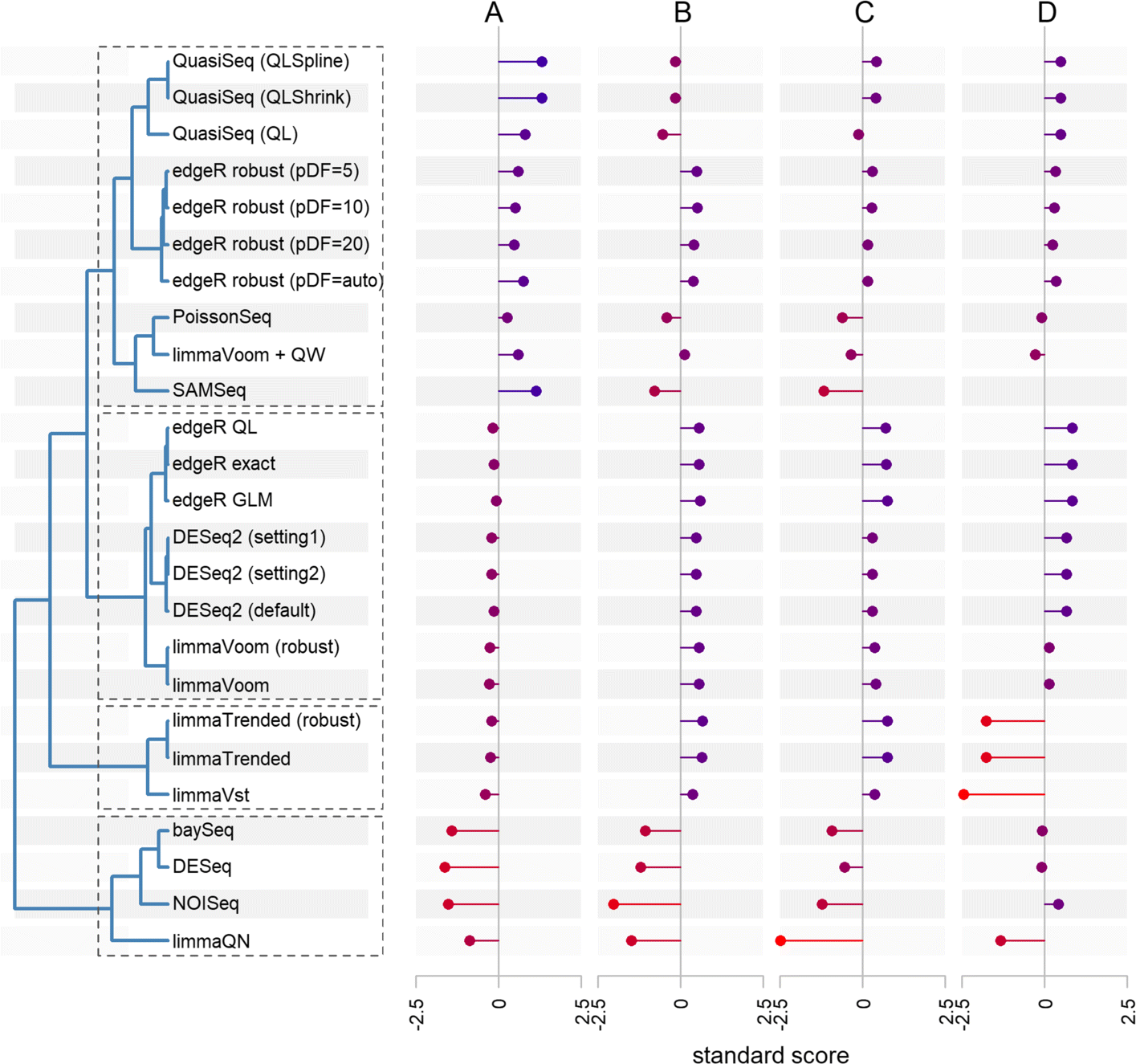
一致性分析
· 分享链接 https://kaopubear.top/blog/2018-12-16-weeklypaper/