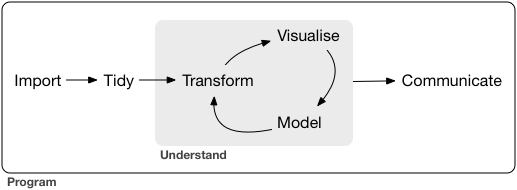
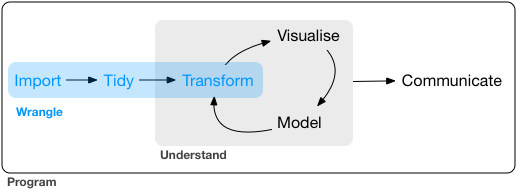
很早之前我一直都是使用命令行来处理软,cut,sed 再 grep 再加上终极 awk 基本上就可以随意的按照需求处理各种软件跑出来的文本文件了,再后来就要开始学习 python,python 学的怎么样了呢?呵呵。 虽然做过统计学课的助教,上课的时候用的也是 R 语言,但是博客里并没有几篇 R 相关的文章。印象中一篇是 给初学者的 R 语言介绍(写这篇文章当时是为了激发一点学生学习 R 语言的热情),另一篇是 R 必备基础知识(写这篇文章当时是因为在写 R 与统计学基础系列文章,就不得不总结一些 R 语言的基础知识)。 说实话,我本身对 R 还是非常喜欢和欣赏的,但是日常的使用需求并不高。就拿最基本的文本处理来说,基本上都可以在 shell 中进行随心所欲地增删改成,处理成很多 R 相关软件需要的文本格式,用需要的 R 包跑一两个命令就可以了。但是目前已经有点厌倦了这样的生活,我后期打算有机会的话用 R shiny 写一点网页工具给大家娱乐,也打算精进一下 R 的画图技能。这里就 have to 用 R 去做一些数据处理,于是就有了这样一系列文章。这篇是关于如何用 R 多快好省的进行数据处理。另外,说句题外话,从我这几天的实际处理数据情况来看,R 有些工作做起来,还是太慢了,也可能是我的业务不够娴熟吧。 Tidyverse 是一系列大名鼎鼎的 data science R 包合集,学会了 Tidyverse 就可以说学会了用 R 进行数据分析,因为它包括的包有 readr, tidyr, dplyr, ggplot2 和 purrr。可以说从读文件到编辑文件再到可视化展示再到编写函数都涉及到了。 数据分析工作主要涉及的内容就是如下几方面的工作。 这篇文章主要涉及输入 (import), 整理 (tidy) 和转换 (transform) 三个部分,这三步合起来也可以称作** data wrangling** 或者 data munging,而正在做这项工作的我们则被称为** data wrangler**。 在 R 中,最基本的文件读取命令是 read.table 和 read.csv。我们这里使用的是更加牛 X 的 readr 包。 readr 解决的关键问题是把 flat file 解析为 tibble。 主要包括如下三个步骤 主要优点 安装及加载命令 reader 支持 7 中文本格式,但是常用的读取命令主要有如下三个 使用实例和方法 多数情况下,如果数据是正规的格式,只需要加入文件路径和名字即可,会根据前 1000 行数据判断数据类型。读取完成后可以使用 为什么要 tidy,因为数据按照一个规矩统后,我们就不用再为数据的各种格式困扰。正所谓,做事之前先立规矩,就是这个道理。 “Happy families are all alike; every unhappy family is unhappy in its own way.” –– Leo Tolstoy “Tidy datasets are all alike, but every messy dataset is messy in its own way.” –– Hadley Wickham 所谓 tidy 的含义其实就是**每一列是一个变量 (variable),每一行是一个观测结果 (observation)。**这个数据格式是后续使用 ggplot 等工具需要的默认格式。 如下图所示: 这里我们要使用的工具是** tidyr**, 是 tidyverse 的几个牛包之一。 用脚想一下,tidy 的数据要求是一个变量在一列,一个观测值在一行,如果我们的数据不满足 tidy 条件,会有什么情况。通常情况下无非是以下两种情况之一。 通常我们会把多个样本的基因表达值构造成为一个矩阵,这时存在的问题就是一个变量分布在不同的几列,每一列是不同的样本名,而并非变量名(变量名应该是基因表达量和样本)。类似于下图有图的问题。 这个时候我们可以使用** gather** 命令进行数据格式转换(数据由宽转长)。 原始数据 spread() 和 gather() 方法类似 原理如下图所示 有些时候我们需要将某一列数据分为两列, 在进行数据分析时,我们通常会对原始的输入数据进行一些处理后再进行下游分析,比如会根据基因的表达量或者基因在不同组间的变异系数对矩阵进行筛选后再进行分析,也可能想要提出某一些有共同特征(同一条染色体的基因)进行后续分析。这些操作都可以通过** dplyr** 方便地完成。 dplyr 主要有以下几个函数,默认情况以下函数会针对整个数据集进行操作,如果使用了 当我打算展开写这部分内容的时候我发现自己实在无法下笔,因为 Rstudio 的 Cheat Sheets 写的实在是太精炼太好,我再整理一遍就是浪费时间了。 dplyr Cheat sheets 下载地址 https://github.com/rstudio/cheatsheets/raw/master/data-transformation.pdf 其他参考资料 http://readr.tidyverse.org/articles/readr.html http://r4ds.had.co.nz/data-import.html#data-import https://github.com/tidyverse/readr 本文作者:思考问题的熊 版权声明:本博客所有文章除特别声明外,均采用 知识共享署名-非商业性使用-禁止演绎 4.0 国际许可协议 (CC BY-NC-ND 4.0) 进行许可。 如果你对这篇文章感兴趣,欢迎通过邮箱订阅我的 「熊言熊语」会员通讯,我将第一时间与你分享肿瘤生物医药领域最新行业研究进展和我的所思所学所想,点此链接即可进行免费订阅。
Tidyverse
优雅地读取数据
install.packages("tidyverse")
library(tidyverse)
read_csv(): comma separated (CSV) filesread_tsv(): tab separated filesread_delim(): general delimited filesread_table(): tabular files where colums are separated by white-space.read_*(file, col_names = TRUE, col_types = NULL,
locale = default_locale(), na = c("", "NA"), quoted_na = TRUE,
quote = "\"", comment = "", trim_ws = TRUE, skip = 0, n_max = Inf,
guess_max = min(1000, n_max), progress = show_progress())

calss()查看一下数据类型,已经是 tbl 格式。data_h3k4 <- read_tsv("46_AB_h3k4_raw.matrix")
# 结果如下
#Parsed with column specification:
#cols(
# peak = col_character(),
# `TAA10-H3K4me3-1-2` = col_integer(),
# `TD265-H3K4me3-1` = col_integer(),
# `Tetra-H3K4me3-1` = col_integer(),
# `TTR13-H3K4me3-1-2` = col_integer(),
# `XX329-H3K4me3-1` = col_integer()
#)
class(data_h3k4)
#结果如下
#[1] "tbl_df" "tbl" "data.frame"
自信地整理数据

library(tidyverse)
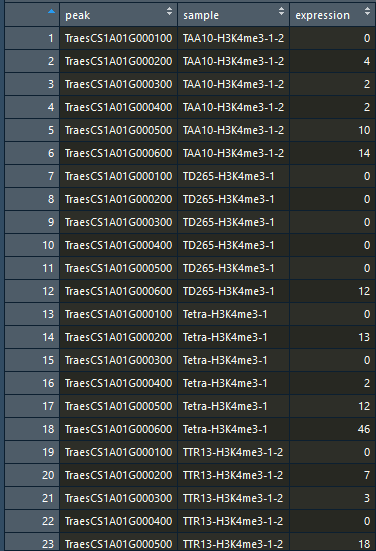
数据由宽变长:gather()

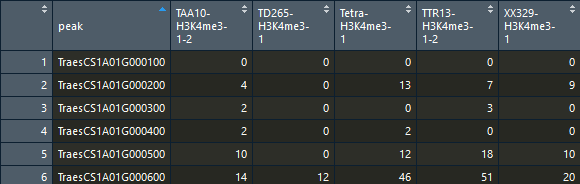
gather(data, key, value, ..., na.rm = FALSE, convert = FALSE, factor_key = FALSE)
#data:需要被转换的宽形表
#key:将原数据框中的所有列赋给一个新变量 key
#value:将原数据框中的所有值赋给一个新变量 value
#…:指定对哪些列进行操作
#na.rm:是否删除缺失值
# 下面的两种写法等效
tmp_tbl <- gather(tmp_data,key = "sample", value = "expression", -peak)
tmp_tbl <- gather(tmp_data,key = "sample", value = "expression", c(2:6))
数据由长变宽 spread()
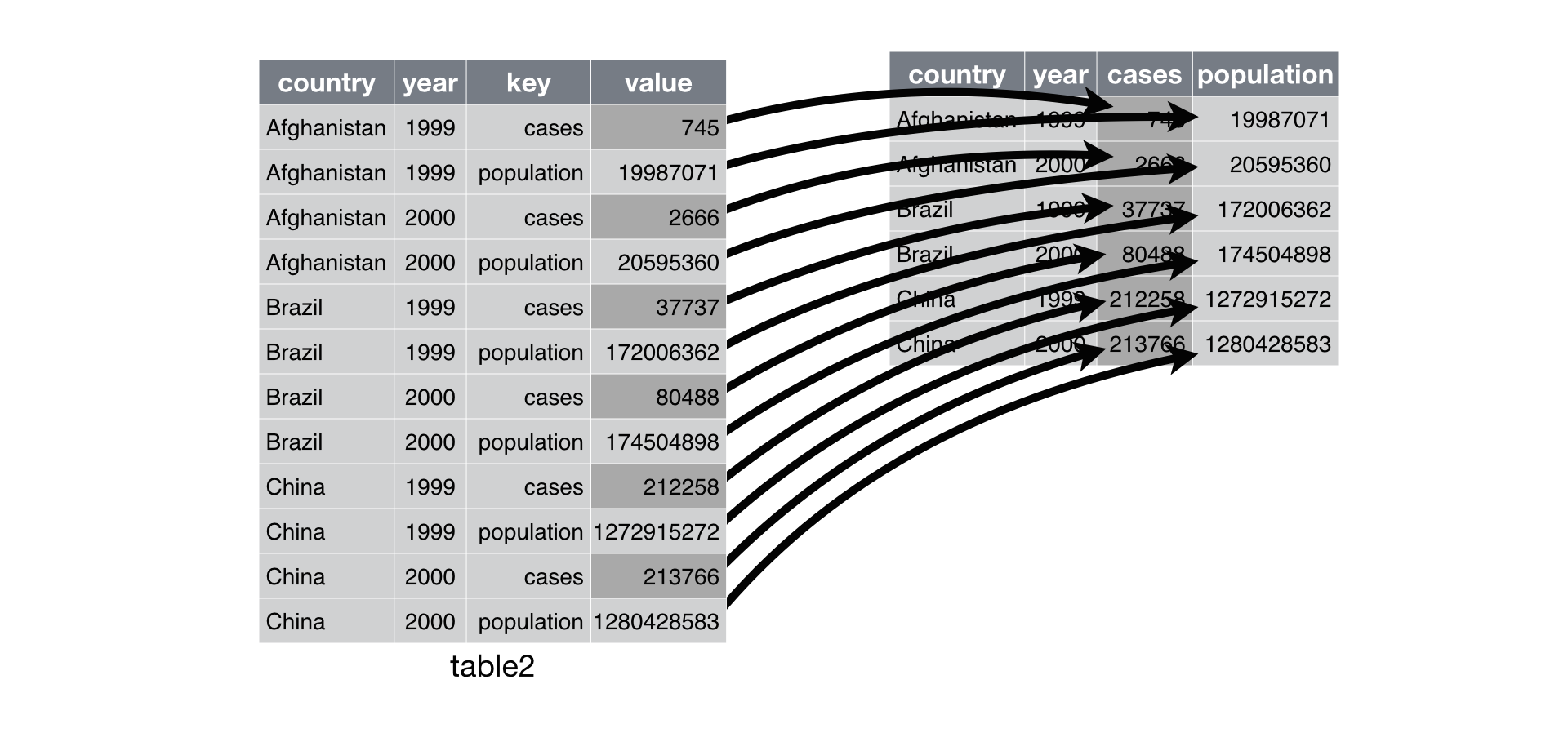
spread(data, key, value, fill = NA, convert = FALSE, drop = TRUE, sep = NULL)
> spread(tmp_tbl,key = "sample", value = "expression")
#结果如下所示
# peak TAA10-H3K4me3-1-2 TD265-H3K4me3-1 Tetra-H3K4me3-1 TTR13-H3K4me3-1-2 XX329-H3K4me3-1
#1 TraesCS1A01G000100 0 0 0 0 0
#2 TraesCS1A01G000200 4 0 13 7 9
#3 TraesCS1A01G000300 2 0 0 3 0
#4 TraesCS1A01G000400 2 0 2 0 0
#5 TraesCS1A01G000500 10 0 12 18 10
#6 TraesCS1A01G000600 14 12 46 51 20
分列:separate
separate(data, col, into, sep = “[^[:alnum:]]+”, remove = TRUE,
convert = FALSE, extra = “warn”, fill = “warn”, …)
#data:为数据框
#col:需要被拆分的列
#into:新建的列名,为字符串向量
#sep:被拆分列的分隔符
#remove:是否删除被分割的列
separate(tmp_tbl, peak, into = c("species","geneid"), sep = "CS")
# species geneid sample expression
#1 Traes 1A01G000100 TAA10-H3K4me3-1-2 0
#2 Traes 1A01G000200 TAA10-H3K4me3-1-2 4
#3 Traes 1A01G000300 TAA10-H3K4me3-1-2 2
#4 Traes 1A01G000400 TAA10-H3K4me3-1-2 2
#5 Traes 1A01G000500 TAA10-H3K4me3-1-2 10
#6 Traes 1A01G000600 TAA10-H3K4me3-1-2 14
#7 Traes 1A01G000100 TD265-H3K4me3-1 0
#8 Traes 1A01G000200 TD265-H3K4me3-1 0
#9 Traes 1A01G000300 TD265-H3K4me3-1 0
列合并:unite
unite(data, col, ..., sep = “_”, remove = TRUE)
#data:为数据框
#col:被组合的新列名称
#...:指定哪些列需要被合并
#sep:组合列之间的连接符(默认下划线)
#remove:是否删除被组合的列
separate(tmp_tbl, peak, into = c("species","geneid"), sep = "CS") %>% unite(peak, c(1:2))
# peak sample expression
#1 Traes_1A01G000100 TAA10-H3K4me3-1-2 0
#2 Traes_1A01G000200 TAA10-H3K4me3-1-2 4
#3 Traes_1A01G000300 TAA10-H3K4me3-1-2 2
#4 Traes_1A01G000400 TAA10-H3K4me3-1-2 2
#5 Traes_1A01G000500 TAA10-H3K4me3-1-2 10
#6 Traes_1A01G000600 TAA10-H3K4me3-1-2 14
tidyr 其它简单命令

从容地进行数据筛选
group_by() 则会分组分别进行操作。
filter()). 筛选行arrange()). 排序行select()). 筛选列mutate()). 增加列summarise()). 汇总计算


· 分享链接 https://kaopubear.top/blog/2018-12-12-rtidyverse/