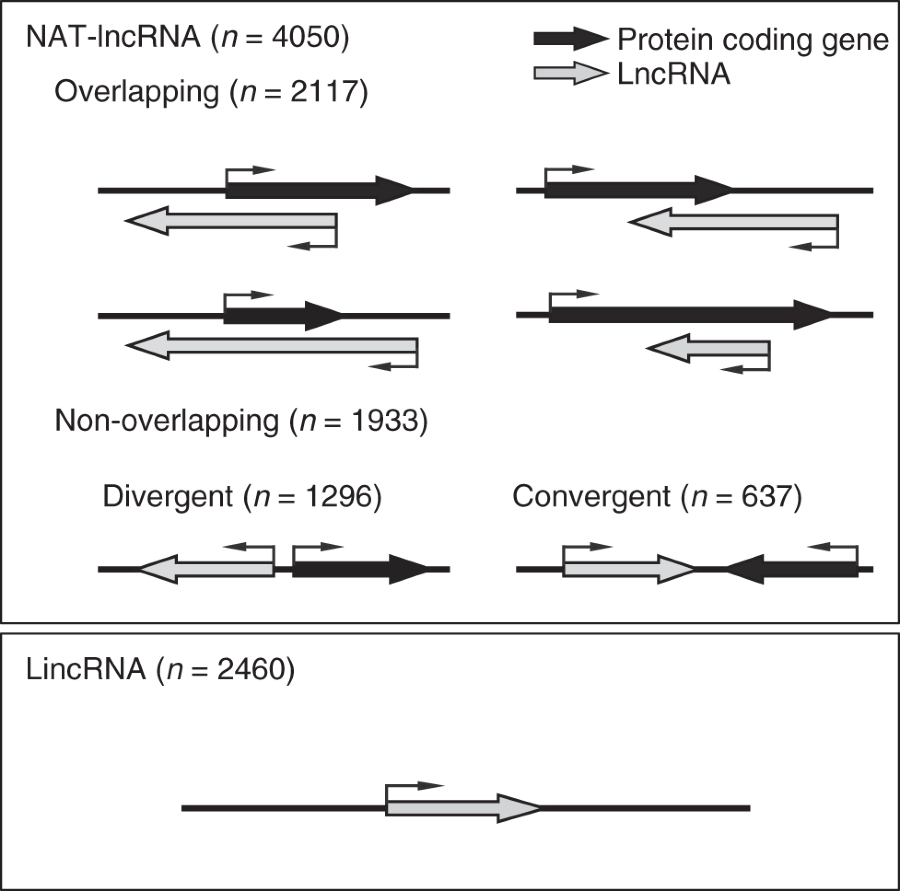
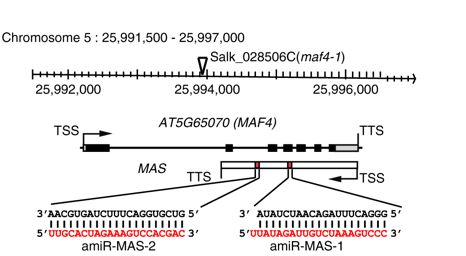
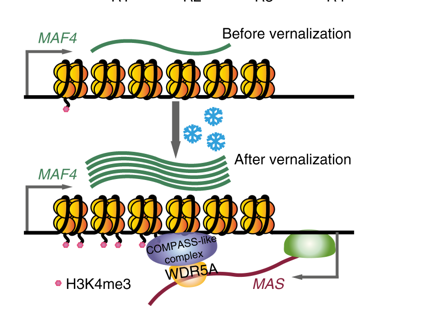
标题:Global identification of Arabidopsis lncRNAs reveals the regulation of MAF4 by a natural antisense RNA DOI(url): 10.1038/s41467-018-07500-7 发表日期:29 November 2018 关键词:lncRNA, H3K4me3, natural antisense transcripts, concordantly expressed 测序和建库类型,作者使用了三种建库方式,分别是去 rRNA, poly(A)+ 和 poly(A)− 。 using high-depth strand-specific RNA sequencing (ssRNA-seq). We generated cDNA libraries for rRNA-depleted total, polyadenylated [poly(A)+] and non-polyadenylated [poly(A)−] RNAs in whole cell extract, nuclear and cytosolic fractions 对转录本进行拼接,从 64,987 genomic loci 找到了 106,421 unique transcripts。 106,421 unique transcripts from 64,987 genomic loci. Among these, 25,245 were previously annotated protein-coding transcripts 去除掉编码 RNA,已知的非编码 RNA,短于 150bp 以及最大 FPKM < 1 的转录本以及编码能力大于 0 的转录本以及和编码 RNA 在同一个方向并且有部分重叠的部分,剩下一共注释了 6510 个 lncRNAs。这里面的数据包括了不同组织和不同胁迫处理。 找到了 lncRNA 之后必然需要进行一些泛泛的统计和分类。首先作者把 NAT lncRNA 分成了 3 类:overlapping (2117), divergent (1296) and convergent (637)。具体每一类的特点如下图所示。 随后作者统计了 lncRNA 特征并和可以翻译的进行了对比,总之就是长度短外显子少 isoforms 也少,当然表达量也低。作者也检查了植物(拟南芥)里的 lncRNA 到底有没有 polyA 尾巴。在我之前的认知里感觉很多 lncRNA 是没有 polyA 的,但是作者的通过对两种建库方式进行严格筛选后发现大部分的 lncRNA 其实是有 polyA 尾巴的。具体信息如下: we found that 1352 lncRNAs were significantly enriched in the poly(A)+ fraction, whereas 198 lncRNAs were significantly enriched in the poly(A)− fraction 我没有想到的一个统计指标是作者对 lncRNA 的核质分布情况进行了分析,发现细胞核比细胞质多的 lncRNA 要多一些。 239 lncRNAs had significantly higher levels in the nuclear fraction than that in the cytosolic fraction, whereas only 43 lncRNAs were more abundant in the cytosolic fraction lncRNA 的表达特异性自然肯定也是要做的,包括不同组织和不同胁迫条件,文章特别提到了也验证出 COOLAIR 冷处理后表达量会升高。 至此都是比较平淡的信息,随后的结论就开始逐渐接触正题。 关于 lncRNA 和 adjacent genes 的关系一直争论,有抑制的也有促进的,各自都有一些实验结果支撑。可能目前的结论就是二者都有。作者计算了他找到的 lncRNA 和对应的基因之间的相关性,发现只要 NAT 相关性系数就要比不是 NAT 的高很多,而且还是正相关。 整个文章中所有的高通量分析数据作者每一步都挑了若干个 RT-qPCR 进行验证。不知道是不是 review 的要求。 We found that the p.c.c. values of overlapping NAT-lncRNA/sense gene pairs were significantly higher than the values between adjacent protein-coding pairs (Fig. 3a), suggesting that overlapping NAT-lncRNAs have a stronger tendency to have positively correlated expression patterns with their sense overlapping genes 正相关还说明不了什么。为了证明是正调控,作者使用 artificial microRNAs knocked down 21 NAT-lncRNAs。 发现,其中 15 个 cognate sense genes 显著下调,3 个上调。 有了现象就要解释结论,目前关于 lncRNA 的作用机制其实已经有不少了,比如当做一个靶点替编码基因吸引敌人,或者通过 R-loop 来发挥作用。当然,如果一个现象用正常的方法很难解释,有一个挺不错的万金油就是用表观遗传学来解释,这里的万金油是个中性词,我自己就是做表观遗传的。 作者想要说明 lncRNA 能够正向调节对应的编码基因,那么最好的方法就是看看是不是和一些类似于 H3K4me3 的激活型修饰联系起来。随后作者很「自然」的 focus 到了一个叫做 MAF4 的基因,对应的 NAT 叫做 MAS,至于为什么要做这个基因也没什么解释。 首先作者用实验证明了这两个转录本到底是编码的调控了非编码的还是反之,然后尝试分析了一些机制,比如会不会双链 RNA 产生的 sRNA,或者影响了编码 RNA 的稳定性。然后通过若干的实验证明了顺式作用 MAS 在转录水平激活 MAF4 表达。 因为已知 H3K4me3 参与了 MAF4 的激活,所以作者就像是不是可以把这个 NAT 和 H3K4me3 联系一下。然后又进行了一系列实验验证。表明确实在 MAS 的突变体中这个表观修饰有变化且一个相关的复合体因子很重要。 从讨论部分可以发现,作者找到的组蛋白 H3K4me3 修饰的 COMPASS-like 复合体核心蛋白组分 WDR5a 之前已经在 Nature 中被报道过和其它的 lncRNA 存在作用,但是找到的机制不同。另外,作者的结果与先前的发现一致,即相邻基因通常具有相关表达,而与其方向无关。 另外,作者也提到了基因之间的“ripple effects ”, 至于原因可能是因为 NAT-lncRNA 的 TSS 与其配对基因之间的平均距离较小,也可能是 NAT-lncRNAs 在激活其配对基因的表达中确实起着至关重要的作用。 作者在计算表达量的时候用的还是 Cuffidff, 我并不是反感 Cuffdiff 但是我看到它就会想到 Tophat2 ,果然在 2018 年作者使用的 mapping 软件还是 TopHat2。转录组拼接用的也是 cufflinks 和 cuffmerge 那一套。 R 的版本是 3.1.0, 这一版 R 的更新日期要追溯到 2014 年 4 月 10 号 了,我猜测要么是这个文章的时间跨度确实已经有三四年了,或者是他们实验室没有一个真正做生物信息且对软件版本有强迫症的人。不过从使用的比对软件来看,也有可能是一个 pipeline 流传了很多年,祖传代码。 相关性计算使用的是 Pearson correlation coefficients,sRNA-seq 用的是 bowtie allowing no mismatches,然后定量单位是 reads per million (RPM)。 RT-qPCR 的时候如何区分正负链 如何保证 amiRNAs 影响反义链的时候不影响正义链的基因表达 关于这个问题,作者在文章中给了正面的回答 Alteration of sense gene expression in amiRNA knockdown lines was not due to targeting of sense genes by amiRNA*s. Eight out of 21 amiRNA*s do not base pair with sense mRNAs at all. The rest of the amiRNA*s have mismatches to corresponding sense mRNAs at critical positions。 Furthermore, most of the amiRNA*s do not have 5’ terminal uridine , making it less likely that they are loaded into the effector AGO1 to suppress gene expression 作者在文章中看似自然的从宏观层面过度到了一个具体的基因,然后讲了一个 NAT 和表观的故事。比较好奇的是其他十几个也找到正向调控的几组为什么都没有提到呢?如果按照正常的套路,一方面可能是他们先做了这个基因然后发现有 lncRNA 可能参与才有了文章开头的高通量试验,另一方面可能是其他的基因也做了,但是似乎表观又解释不了。 文章中关于 ChIP 的部分并没有做高通量试验,或者做了至少没有放在文章中,只是用 ChIP-qPCR 去验证。只要做一个 ChIP-seq 的实验很容易看看其他十几个基因的修饰情况,甚至可以 K27, K36 这些修饰都做一做。如果能有一半的基因做出表观的相关性,我都会更相信这个机制。 nature 系列的杂志目前似乎可以公开作者和文章 review 之间的质疑和回应内容,在这篇文章的附件中可以看到完整的 review 意见,其中「如何保证 amiRNAs 影响反义链的时候不影响正义链的基因表达」也是 review 的问题。 个人感觉仔细阅读 review 和 作者之间沟通的这个记录比阅读原文更有意思。 植物中研究的比较多的几个 lncRNA COLDAIR: Swiezewski, S., Liu, F., Magusin, A. & Dean, C. Cold-induced silencing by long antisense transcripts of an Arabidopsis Polycomb target. Nature 462, 799–802 (2009). APOLO: Ariel, F. et al. Noncoding transcription by alternative RNA polymerases dynamically regulates an auxin-driven chromatin loop. Mol. Cell 55, 383–396 (2014). ELENA: Seo, J. S. et al. ELF18-INDUCED LONG-NONCODING RNA associates with mediator to enhance expression of innate immune response genes in Arabidopsis. Plant Cell 29, 1024–1038 (2017). 本文作者:思考问题的熊 版权声明:本博客所有文章除特别声明外,均采用 知识共享署名-非商业性使用-禁止演绎 4.0 国际许可协议 (CC BY-NC-ND 4.0) 进行许可。 如果你对这篇文章感兴趣,欢迎通过邮箱订阅我的 「熊言熊语」会员通讯,我将第一时间与你分享肿瘤生物医药领域最新行业研究进展和我的所思所学所想,点此链接即可进行免费订阅。文献信息
文献概述
笔记
文章主要内容和结论
讨论部分几点信息
主要方法
比较关心的几个问题
review 怎么看
相关文献
· 分享链接 https://kaopubear.top/blog/2018-11-30-weeklypaper/