最近读到一篇 博文,介绍了学习 RNAseq 数据分析非常推荐的两篇综述和七个在线教程并发表了相关评论。本文将进行简要整理并对相关评论内容进行大致翻译,供国内读者参考。 这篇文献 基于实际 qRT-PCR 测量得到的结果对各种统计分析技术进行了详细的比较。他们得出的主要结论是 NOIseq,limma + voom 和 DESeq2 是精度,准确度和灵敏度三者平衡最好的几组软件。本文的图 1 展示了 RNAseq 数据分析不同阶段中使用的各种工具的精彩总结。 2016 年的这篇文献应该叫做 RNA-seq 分析方法中的沃尔玛. 它方方面面都有涉及到不少东西因此也被很多文章引用。 接下来将会提到七个详细的在线教程。你可以下载他们的数据然后尝试使用 R 重现分析步骤。唯一的缺点是这些教程主要介绍了六七年前发布的成熟方法。如果你正在找过去两三年内发布的那些方法(例如 Kallisto,Salmon)就需要查阅它们的用户手册了。 这个教程 在统计方面非常强,你可以下载他们编写的很好的 pdf 版本 教程。 这个教程 是生物信息教程中为数不多带有详细实验步骤的。 来自加州大学河滨分校的 Thomas Girke 的教程 非常优秀, 这里 还有一个他旧版教程的 PPT。 另外,来自同一个学校的 Rakesh Kaundal 也有一个 详细的教程。 Mike Love 是 DESeq2 的主要作者,他在 bioconductor 上有一个 很棒的教程. 同时别忘了看 DEseq2 的 vignette. 这个小组发表了和各种 Bioconductor 主题相关的 一系列详细教程 其中就包括是用 edgeR 来分析 RNAseq。 这个教程 是一个而来自剑桥大学的优秀在线教程。 Weijun Luo 是 GAGE 和 pathview 这两个工具的作者,他发表过一个关于 RNAseq 分析流程的 简要总结。 上述所有提到的 PDF 相关文档已经下载打包,下载地址:链接:https://share.weiyun.com/5rvQy3e 本文作者:思考问题的熊 版权声明:本博客所有文章除特别声明外,均采用 知识共享署名-非商业性使用-禁止演绎 4.0 国际许可协议 (CC BY-NC-ND 4.0) 进行许可。 如果你对这篇文章感兴趣,欢迎通过邮箱订阅我的 「熊言熊语」会员通讯,我将第一时间与你分享肿瘤生物医药领域最新行业研究进展和我的所思所学所想,点此链接即可进行免费订阅。两篇综述
J. Costa-Silva et al.
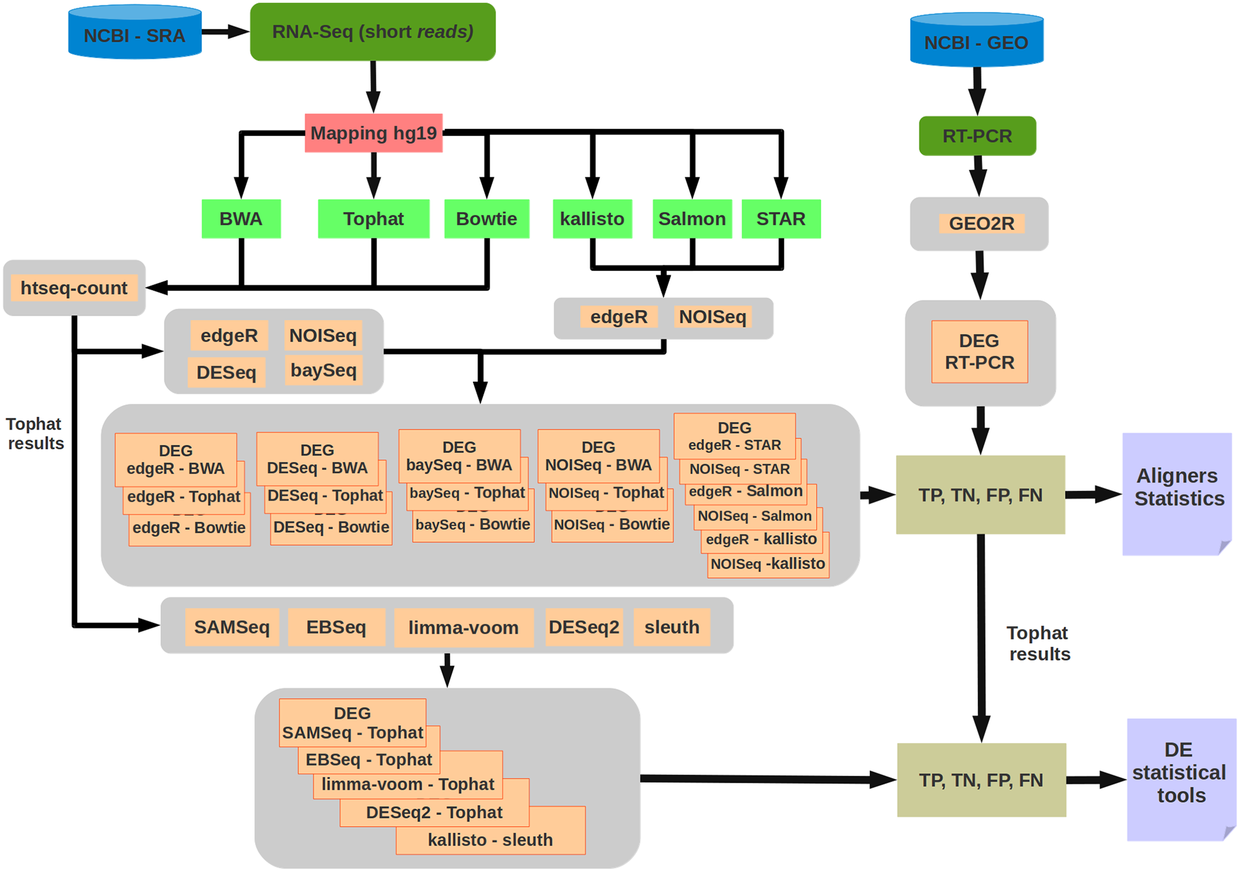
Ana Conseca et al.
在线教程
Annick Moisan, Ignacio Gonzales, Nathalie Villa-Vialaneix
Friederike Dündar, Luce Skrabanek, Paul Zumbo
Thomas Girke, Rakesh Kaundal (UC Riverside)
Mike Love
Stanford Bios221
University of Cambridge
List of All Analysis Steps including Biological Analysis
· 分享链接 https://kaopubear.top/blog/2019-02-23-rnaseqreviewandcourse/